金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
从《The Scientist》2019年度具有代表性和突破性的生物技术来抛个砖:
一、人工智能技术
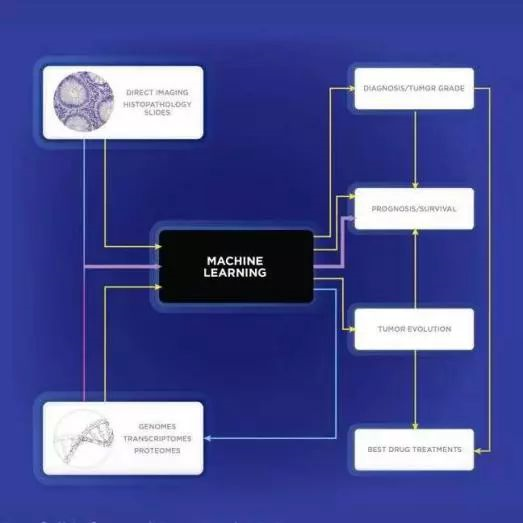
纵观今年生物学的许多重大发现,机器学习作为一种工具不断促进生命科学领域的发展。
今年的众多研究项目中,有研究人员报道他们已成功通过机器学习来检查机体是否存在癌症或病原体感染;也有研究人员通过机器学习鉴定血液样本中与糖尿病患者血管并发症相关的表观遗传标志物……
二、活细胞记录技术

今年8月,麻省理工学院的研究人员报道了利用DNA以及类似CRISPR的碱基编辑机制记录细胞内部的生命活动,然后通过测序对其进行解码的方法。他们对大肠杆菌和人类细胞进行了工程改造,使它们可以记录多种分子事件以及它们发生的时间和顺序。
该方法的基本操作单位是两种类型的基因序列:一种在诱导型启动子的控制下编码特定的gRNA,一种编码与核酸酶的CRISPR-Cas9酶(与gRNA结合)融合的碱基编辑酶。遗传序列被转染到细胞中,当这些细胞暴露于诱导剂(诱导剂是实验人员希望在DNA中蚀刻的任何分子事件)时,表达gRNA序列,RNA结合到Cas9-base编辑器融合蛋白,然后将其转运到目标DNA序列(与gRNA匹配)进行编辑。这些编辑可以稍后通过DNA测序进行分析。文章的共同作者,麻省理工学院的Timothy Lu说,这项研究在环境毒素的检测和发育过程的记录方面也具有潜在的应用价值。【1】
三、DNA芯片技术
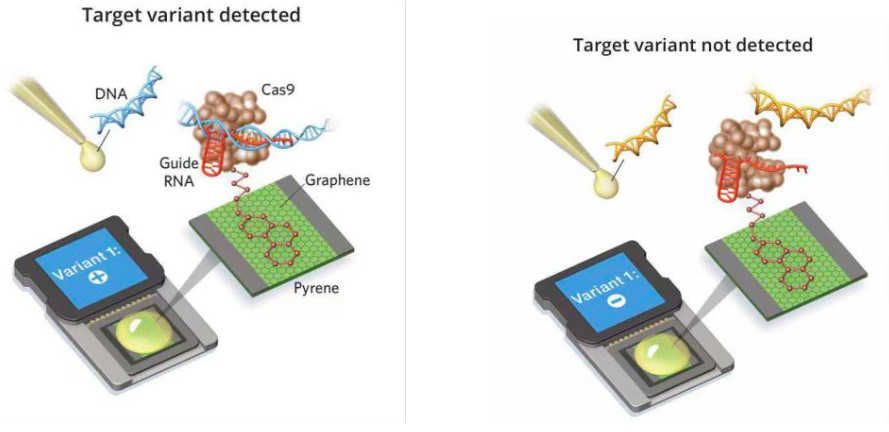
今年,基于CRISPR-Cas9编辑进行的另一项创造性技术是开发了针对特定DNA序列的检测设备。目前,在众多实验室中主要采用目标扩增的方式,在DNA样本中寻找特定的碱基序列。但这种方法需要耗费时间,还有可能产生偏差。
为了避免目标扩增的缺点,加利福尼亚州Keck Graduate 研究院Kiana Arand的团队将研究对象转向了CRISPR-Cas核酸酶家族,当它与特定的指导RNA配对时,可以搜寻整个基因组以发现并切割精确的序列。他们将Cas9酶与RNA和石墨烯芯片结合,且经过改造设计后不会切割DNA。当RNA-Cas9复合物与其目标DNA序列连接,芯片内的电场就会发生变化,继而可进行阳性读数。
四、PE编辑技术
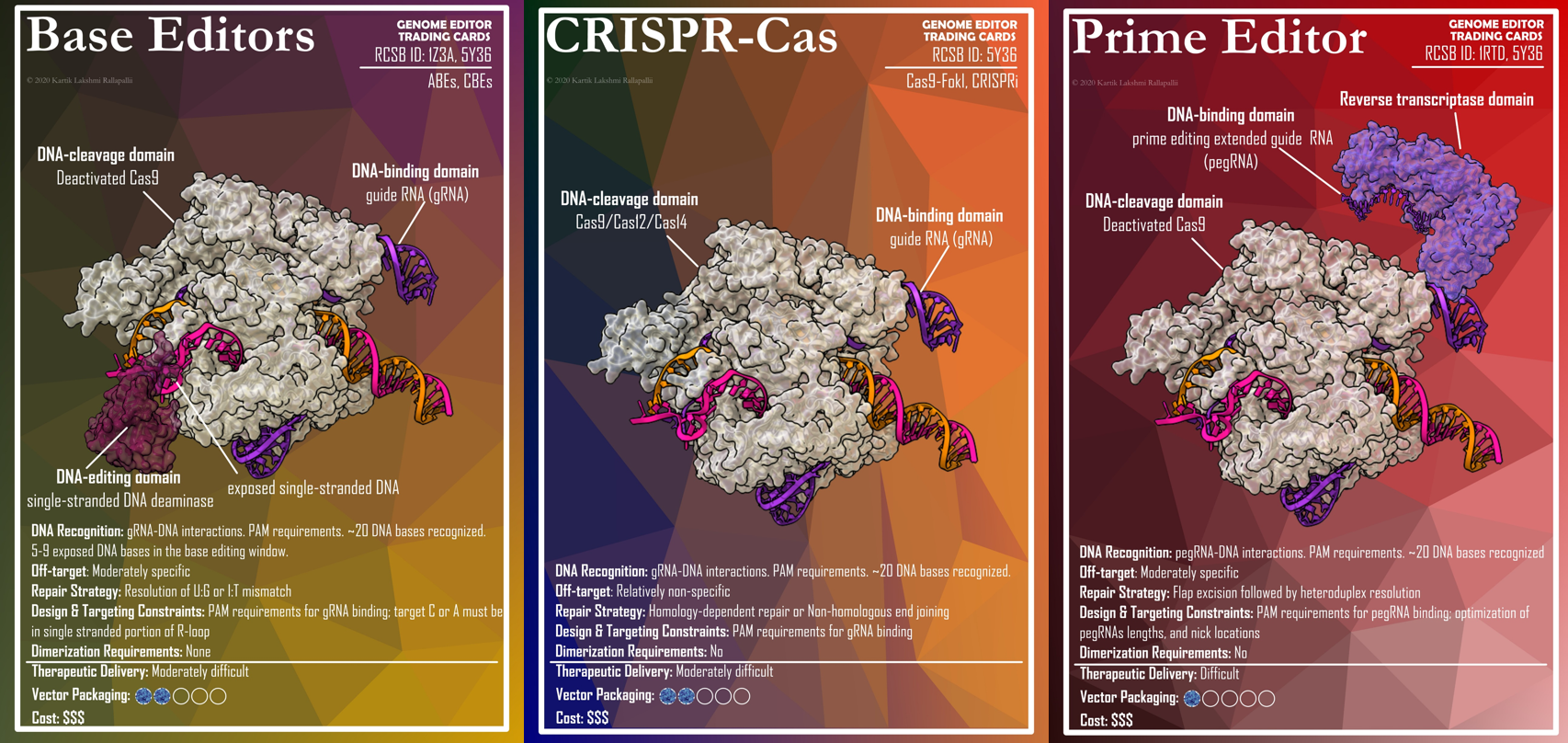
David Liu及其团队研究的编辑技术prime editing,旨在通过避免双链DNA断裂来降低CRISPR的脱靶效应并提升CRISPR的准确性,该技术在今年10月于Nature发表。PE编辑技术仅通过切割一条DNA单链添加、去除或取代碱基对,即可改写DNA。
它使用的是CRISPR系统中常用的Cas9核酸酶,但将该酶与一种名为pegRNA的引导RNA和另外一种逆转录酶相结合,后者可以向基因组中添加新的序列或碱基。一旦新的遗传物质掺入到DNA的切割链中,PE就会对未编辑的DNA链进行刻迹,向细胞发出信号对它进行重建,并与已编辑的链进行匹配【2】。详见BioArt报道:
BioArt生物艺术:专家点评Nature | David Liu再出重磅基因编辑新工具,可实现碱基随意转换与增删
五、新型多功能干细胞诱导技术

Shinya Yamanaka在2006年首次发表了诱导多功能干细胞的方法,该方法通过在分化过的细胞中过表达Oct4,Sox2,Klf4,cMyc四种转录因子,将细胞重置为多能状态,形成诱导多能干细胞(iPSC)。且Oct4是四个过表达的转录因子中最重要的一个。但在今年十一月,马克斯·普朗克分子生物医学研究所 (Max Planck Institute for Molecular Biomedicine) 的Sergiy Velychko团队宣布,他们不仅能在不调整Oct4水平的情况下制造出小鼠iPSC,而且具有更高的效率。Yamanaka在给《The Scientist》的邮件中谈道:“若这种新的方法可应用于人的细胞,将极有利于iPS细胞在临床上的应用。”【3】
参考文献
[1] F.Farzadfard et al.,“Single-nucleotide-resolution computing and memory in living cells,” Molecular Cell ,75:P769-780.E4,2019.
[2]A.Anzalone et al.,“Search-and-replace genome editing without double-strand breaks or donor DNA,”Nature,doi:10.1038/s41586-019-1711-4,2019
[3]S. Velychko et al.,“Excluding Oct4 from Yamanaka cocktail unleashes the developmental potential of iPSCs,” Cell Stem Cell, doi:10.1016/j.stem.2019.10.002, 2019. |
|
 /3
/3 