金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
CRISPR筛选是一种革命性的高通量功能基因组学技术,它将强大的CRISPR-Cas基因编辑工具与大规模并行筛选策略相结合。在实验中,研究者将包含数万至数百万条sgRNA的病毒文库导入细胞群体,使单个细胞通常仅携带一种sgRNA,从而靶向特定基因。随后,细胞群体被置于特定选择压力下(如药物处理、病毒感染、特定生长条件或基于增殖、死亡、荧光标记等表型的分选)。经过筛选,那些sgRNA(及其靶向基因)在特定条件下赋予细胞生存优势或劣势的细胞群会被富集或耗竭。
通过深度测序分析筛选前后细胞群体中sgRNA的丰度变化,研究人员能够大规模、无偏倚地鉴定在特定生物学过程(如细胞存活、药物敏感性/耐药性、病毒感染、信号通路、癌症转移等)中发挥关键作用的基因。高通量测序结果的可视化是CRISPR筛选研究的核心环节,其呈现方式因算法和筛选指标的选择而略有差异。本文将介绍几种常用的CRISPR筛选结果可视化方法。
<hr/>
01 RRA Score排序图
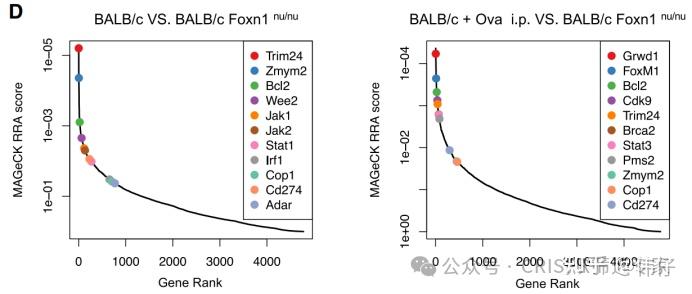
MAGeCK RRA算法输出的RRA score越小,表明基因在正向(positive)或负向(negative)筛选中富集或耗竭越显著。研究者常将RRA score排序后的Top基因(如Top 10)作为候选基因展示。示例:图1展示了RRA score排序后的Top基因 [Wang et al., Cell, 2021]。
<hr/>02 随机散点图
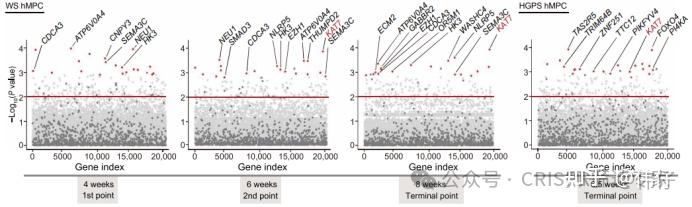
MAGeCK RRA算法会为正向和负向筛选分别提供RRA score、p值、FDR值和LFC值(其中LFC值相同)。可以选用-log10(p-value)、-log10(FDR)或RRA score等指标作为纵坐标,对所有基因绘制随机散点图。显著变化的基因会分布在散点图的顶部。
示例:图2展示了以-log10(RRA p-value)为纵坐标的随机散点图 [Wang et al., Sci Transl Med.,2021]。
<hr/>03 基于差异值的Rank图
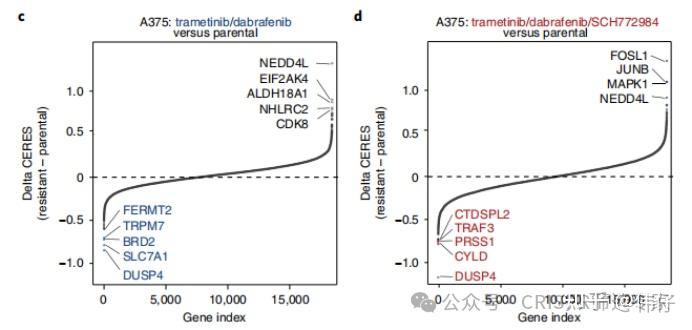
与RRA score(范围0-1)不同,如果筛选参数数值呈对称分布(如MAGeCK RRA输出的LFC值或MAGeCK MLE算法提供的差异β值(实验组 - 对照组)),则可以绘制双向排序图(Rank图)。这种图能直观展示正向和负向筛选中均显著变化的基因。示例:图3展示了基于差异CERES值绘制的Rank图 [Takahiro et al., Nat Genet.,2021]。
<hr/>04 火山图
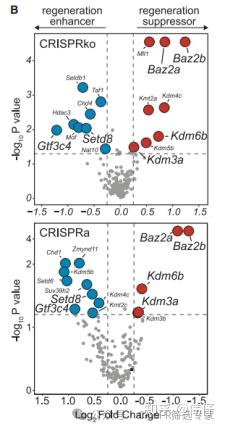
火山图是展示差异分析结果的常用方法。利用MAGeCK RRA提供的LFC值和p值(需注意:同一基因有正向p值pos.p和负向p值neg.p),可将LFC > 0的基因使用pos.p,LFC < 0的基因使用neg.p绘制火山图。图中横轴通常代表LFC,纵轴代表-log10(p-value),能同时清晰展示正向和负向筛选中的Top Hits。
示例:图4展示了火山图在CRISPR筛选Top基因中的应用 [Jia et al., Cell Stem Cell. 2022]。
05 九宫格图
这种图常用于比较不同实验条件下的筛选结果(如不同药物浓度、不同时间点)。基于MAGeCK MLE算法输出的β值,可以设定阈值将基因分为不同类别(如只在条件A显著、只在条件B显著、在两者均显著等),并展示在九宫格中。
示例:图5展示了基于MAGeCK MLE β值绘制的九宫格图 [Feng et al., Protein Cell,2022]。

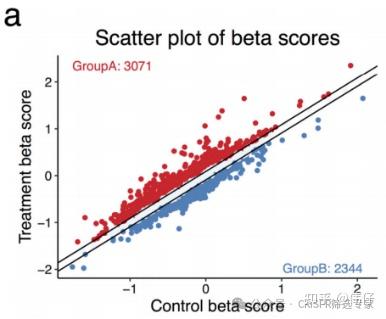
<hr/>06 对角线火山图
这是一种特殊形式的火山图,尤其适用于展示对照组与实验组的比较结果。它基于MAGeCK MLE的β score,使用CutoffCalling函数(例如设定scale_cutoff=2)自动确定正向和负向筛选的阈值。结果以对角线划分区域,红色区域的基因敲除后倾向于耐药(或耐受筛选压力),蓝色区域的基因敲除后则对压力更敏感。示例:图6展示了对角线火山图 [Wang et al., Nat Protoc.,2019]。
<hr/>07 sgRNA分布图
针对感兴趣的Top基因,研究者常展示其靶向sgRNA的变化情况。以每个sgRNA的LFC值为依据,绘制每个基因所有靶向sgRNA的LFC分布(如小提琴图、点图或条带图)。sgRNA的表现趋势越一致(如所有sgRNA的LFC均显著为正或负),表明该基因的功能效应越可靠。示例:图7C展示了特定基因的sgRNA LFC分布情况 [Cortez et al., Nature,2020]。
<hr/>研美生物致力于提供CRISPR筛选的全流程服务,涵盖文库设计与构建、病毒包装、文库细胞制备、筛选定制及NGS数据分析。凭借丰富的项目经验,我们已为众多高校、科研院所及医院提供专业支持。为满足科研工作者对CRISPR筛选结果多样化和深度可视化的需求,我们现推出进阶版CRISPR NGS分析服务报告(私信领取报告详情)。该报告包含上述多种高级可视化结果及深度解读。Demo数据来源: Wei W, Geer MJ, Guo X, Dolgalev I, Sanjana NE, Neel BG. Genome-wide CRISPR/Cas9 screens reveal shared and cell-specific mechanisms of resistance to SHP2 inhibition. J Exp Med. 2023 May 1;220(5):e20221563. doi: 10.1084/jem.20221563. Epub 2023 Feb 23. PMID: 36820830; PMCID: PMC9998968.
原文地址:https://zhuanlan.zhihu.com/p/1919799851929040377 |
|
 /3
/3 